通过操纵DNA以解决数学问题正在使“计算”的含义被重新界定。一看到“计算机”这几个字,人们立刻就会想起键盘和监视器的情景,诸如“只读存储器”(R0M)、“随机存取存储器”(RAM)、“千兆字节”和“兆字节”等等之类的术语也会同时涌人脑海中。我们已经习惯了这样一个概念:计算是通过硅片上的电子元件进行的。
但是计算非得是采取这样一种方式不可吗?你在读这篇文章时所用的计算机与PC微机相比几乎没有一点相似之处。或许我们对计算的看法过于狭隘了。如果计算机无处不在,而且能够表现为多种形式,情况又如何呢?是否可能存在一种由相互作用的分子进行计算的液体计算机呢?答案是肯定的。下面就是关于DNA计算机的故事。

重新发现生物学
我是在1993年当我首次走进一家分子生物实验室时开始涉足于这个故事的。虽然我的专业是数学和计算机科学,但我也曾搞过一点艾滋病研究,我当时认为,现在也仍然认为它具有重要意义[参看《科学》1993年9月号“科学与大众”专栏John Rennie所著的“平衡的免疫性”一文]。遗憾的是,我始终没有能让艾滋病研究界了解我的设想,这真是太出乎意料了。因此,为了使自己能成为一个更让人信服的宣传者,我决意要深化自己对HIV生物学的了解。这样我就进了那家分子生物学实验室。在该实验室里,我得到Nickolas Chelyapov(现在是我自己的实验室的首席科学家)的指导,开始学习现代生物学的方法。

我很快就着了迷。通过我自己的双手,我创造出了自然界不存在的DNA。我把该DNA引入了细菌中,在细菌体内它起着生产蛋白质的蓝图的作用,生产出将会改变细菌本身特性的蛋白质。
在这段紧张学习的时期中,我开始阅读《基因的分子生物学》(The Molecular Biology of the Gene)这本经典著作,它的作者之一是以Watson—Crick的声名著称于世的James D.Watson(Watsons与Francis Harry Cornpton Crick共同发现了DNA的双螺旋结构)。我对生物学的看法开始发生根本性的变化。生物学不再是研究那些在冰箱中散发出古怪气味的东西的科学了(这是我从六十年代起在伯克利加利福尼亚大学读书时就形成的观点)。它正在经历一场革命,并正在迅速地获得先前人们认为只有物理科学才具备的那种深度和力量。生物学现在成了研究存储在DNA中的信息——代表腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶的4个字母A、T、G和C组成的字母串——以及研究信息在细胞中发生的变化的学科。而这里数学是有用武之地的!
一天晚上很晚的时候,当我躺在床上读Watson的著作时,看到了介绍DNA聚合酶的一段文字。DNA聚合酶是酶中之王,是生命的制造者。在合适的条件下,有了一股DNA,DNA聚合酶便产生出第二条互补的“Watson—Crick股,在这一DNA股中每个C用一个G代替,每个G用一个C代替,每个A用一个T代替,而每个T用一个A代替。例如,给定一个具有CATGTC序列的分子,DNA聚合酶就将产生一个其顺序为GTACAG的新分子。聚合酶使DNA能够复制,而这又进一步使细胞能够复制,最终使你能够复制。”对于一位严格的简化论者来说,用DNA聚合酶来复制DNA就是生命的全新奥秘之所在。
DNA聚合酶只有一个分子的神奇的纳米机器,它“跳”到一股DNA上并沿着它滑下去,“读出”它经过的每一个碱基,并把其互补碱基“写到”一条新的、正在生长的DNA股上。当我躺在床上对这种神奇的酶钦佩得五体投地之时,突然发觉这种酶与著名的英国数学家Alan M.Turing在1936年描述的某种东西有相似之处。当时Turing已经开始对“可计算性(computability)这一概念进行严密的研究(Kurt Gdel、Alonzo Church和S. C.Kleene也各自独立地在进行此项研究)。这一纯理论的研究先于真正的计算机问世大约1O年左右,并产生了二十世纪的若干重大数学成就[见《科学美国人》1971年3月号Howard Delong所著“算术中的未解决问题”(Unsolved Problems in Arithmetic)-文,以及《科学》1988年11月号Gregory J. Chaitin所著“算术中的随机性一文。]

为了进行他的研究,Turing发明了一种“玩具”计算机,现在称为Turing机。这一装置并不是真正用来计算的,而只是适合于数学研究用的概念上的计算机。为此它必须极其简单——而Turing也非常高明地做到了这一点。Turing机的一种型式由两条纸带和一个称为“有限控制”的装置构成,该装置沿着“输入”纸带移动并读出数据,同时沿着“输出”纸带移动并读出和写人其它数据。有限控制可以用非常简单的指令来编程,人们很容易编写一个程序来读出输人纸带上的一串A、T、C和G,。然后把互补的Watson—Crick串写在输出带上。Turing机与DNA聚合酶之间的相似性实在是再明显不过了。
但是有一项重要的信息使得这种相似性真正地引人注目:Turing的玩具计算机被证明是万能的——尽管看起来非常简单,但它却可以在编程后计算任何能够被计算的问题。(这一命题基本上就是著名的“Church论点”的内容。)换言之,可以对一台Turing机编程使之产生Watson—Crick互补串、分解数字、下象棋,等等。意识到这一点后,我一下子从床上坐起来,对我的太太Lori说:“Jeez,那些东西能够计算。”那一夜余下的时问里我没有睡觉,一直在冥思苦想如何找出一条用DNA来解题的途径。
我最初的设想是依照Turing机的样子来做出一台DNA计算机,其有限控制用酶来代替。值得注意的是,几乎10年以前,IBM公司的Charles H.Bennet和Rolf Landauer提出了大致相同的设想[见《科学》1985年第11期“计算的基本物理极限”一文]。遗憾的是,虽然已知有一种酶(DNA聚合酶)可以作出Watson—Crick互补串,但看来不可能存在任何一种酶可以执行其它的重要任务,如分解数字等。
这就暴露了关于生物技术专家的一个重要事实:我们实际上是一群贼。我们从细胞那里偷东西。我们还远远不能从头创造诸如DNA聚合酶之类的神奇的分子机器。幸运的是,30亿到40亿年的进化过程已经产生了充满各种神奇的小机器的细胞。正是这些从细胞那里偷来的机器使现代生物技术有可能实现。然而,能下象棋的分子机器显然从未进化出来。因此,如果我要建造一台能够做些有意义的事情的DNA计算机,我就必须用手头现有的工具来完成这一工作。这些工具基本上有如下一些:
1、Watson—Crick配对。如前所述,每一股DNA都有它的Watson—Crick互补股。凑巧的是,如果溶液中的一个DNA分子遇到了它的Watson—Crick互补分子,那么这两股DNA就将“退火(annea1)——也就是彼此卷绕在一起,形成有名的双螺旋结构。这两股DNA不是通过共价键结合,而是借助于较弱的力(如氢键)结合在一起。如果溶液中的一个DNA分子遇到了另一个彼此并不互补的DNA分子(并且没有较长的互补段),那么这两个分子将不会退火。
2、聚合酶。聚合酶把信息从一个分子拷贝到另一个分子上。例如,DNA聚合酶将根据一个DNA模板制造出一个Watson—Crick互补DNA股。事实上,DNA聚合酶需要一个“启动信号”告诉它在何处开始进行互补拷贝。此信号由一个“启动子”(Primer)提供。启动子是一个(可能相当短的)DNA片断,它通过Watson—Crick互补性结合到模板上。一旦发现了这样一个启动子一模板对,DNA聚合酶就开始向启动子添加碱基,以作出模板的一个互补拷贝。
3、连接酶。连接酶把分子结合在一起。例如,DNA连接酶将使两股DNA靠近,并用共价键把它们结合成单一的股。细胞利用DNA连接酶来修复DNA股的断裂(例如在皮肤细胞受到紫外光照射后所发生的DNA股的断裂)。
4、核酸酶。核酸酶把核酸切断。例如,限制性核酸内切酶将“搜寻”一股DNA上的一个预先确定的碱基序列,并且在发现该序列后,就把这个分子切为两个片断。EcoRI(自大肠埃希氏菌EScheriChiaColi)是一种限制性酶,它将在序列GAATTC的“G”之后把DNA切断——它几乎从不在其它任何一个部位上切断一股DNA。有人认为,限制性酶是进化出来保护细菌免受病毒之害的(不错,甚至细菌都有病毒!)例如,大肠埃希氏菌有一种手段(甲基化)保护它自己的DNA免遭EcoRI切断,但是一个带有致命的GAATTC序列的入侵病毒将被切成碎块。我的DNA计算机没有使用限制性酶,但是这些酶已被其它许多研究小组用于随后进行的实验中。
5、凝胶电泳。这一技术不是从细胞那里偷来的。含有各种不同的DNA分子的溶液被置于一块凝胶板的一端,同时向溶液施以电流。带负电的DNA分子将向阳极移动,而较短的DNA股将比较长的DNA股移动得快。因此,这一过程把DNA按长度的不同分离开来。使用特殊的化学物质和紫外光,可以看到各种不同长度的DNA分子停下来以后在凝胶中形成的条带。
6、DNA合成。现在可以把一个DNA序列写在一张纸上,把它寄给一家商业合成机构,几天内就可以收到一只试管,其中装有大约10DNA分子,所有这些分子(或至少是大部分分子)都具有规定的序列。现在长度为100左右的序列能够可靠地通过这种方法制备。对于长度为20的序列,其费用约为25美元。这些分子将装在一个小试管中以干的形式交付给用户,其外观像一个无定形的小的白色团块。
以上这些手段看来都不可能帮你下象棋,但是三十年代的大逻辑学家们还教给了我们另外一个重要事实:计算是很简单的。为了建造一台计算机,实际上只需要两样东西,一是存储信息的方法,二是作用于该信息的若干简单的运算。Turing机把信息作为字母串存储在纸带上,同时用有限控制中的简单指令来处理纸带上的信息。电子计算机则把信息以0和1的数字串的形式存储在存储器中,同时用处理芯片上所提供的操作对数据进行处理。值得注意的是,几乎任何一种存储信息的方法和任何一组作用于信息的运算都是完全适用的。
适用于做什么呢?适用于进行一般的计算——即计算任何能够被计算的东西。为了使你的计算机作出Watson—Crick互补物或者是下象棋,你只需要从正确的输入信息着手并使用一系列正确的运算——也就是运行一个程序。DNA是存储信息的一种极好手段。事实上,几十亿年来细胞一直在用这种方法存储“生命蓝图”。此外,像聚合酶与连接酶之类的酶则一直被用于对这一信息进行操作。建造一台万能计算机所需要的东西是否已经够了呢?由于有了三十年代的经验,我确信这个问题的答案是肯定的。
哈密顿路径问题
下一项任务就是选择一个问题来解决。这个问题不应当给人一种是故意编造出来以迎合计算机的印象,而且它应当能证明这一新颖计算方法的潜力。我选择的问题是哈密顿路径问题(Hamiltonian Path Problem)。
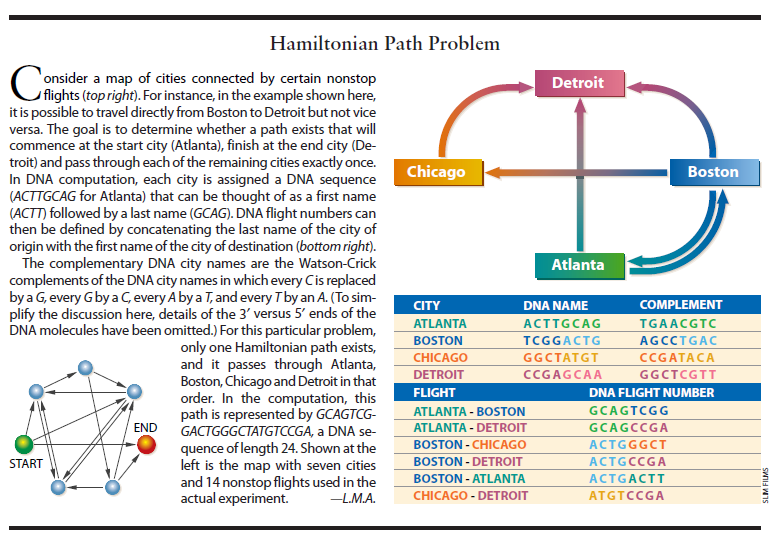
William Rowan Hamilton是十九世纪中期爱尔兰的皇家天文学家。用箭头(有向边)表示地图(网络)中的各城市(顶点)之间的直达航班例如,你可以从波士顿乘直达航班到芝加哥,但不能从芝加哥乘直达航班到波士顿。你的任务(即哈密顿路径问题)就是要确定,是否存在着这样一系列的互相衔接的航班(即一条路径),它从亚特兰大(起始顶点)出发,结束于底特律(终止顶点),并且经过其它每个城市(即波士顿和芝加哥)恰好一次。这样一条路径称为哈密顿路径。在图2所示的例子中,很容易看出存在一条唯一的哈密顿路径,它依次经过亚特兰大、波士顿、芝加哥和底特律这几个城市。如果把起点城市改为芝加哥,而终点城市改为亚特兰大,则很显然将不存在哈密顿路径。
一般地说,给定一个由有向边组成的网络,并规定一个起始顶点和一个终止顶点,那么,当且仅当存在着一条从起始顶点出发,并在终止顶点结束、并且经过其它每个顶点恰好一次的路径时,我们说该网络存在哈密顿路径。哈密顿路径问题就是对于任意一个规定了起始顶点和终止顶点的给定网络,确定它是否存在一条哈密顿路径。计算机科学家们已经广泛研究过哈密顿路径问题,但从未找到一种有效的(即快速的)算法来解决这个问题事实上,看来情况有可能是,即使我们运用目前世界上最好的算法和计算机,对于某些顶点数不到100的网络,要确定其是否存在哈密顿路径也将需要几百年的时间。
七十年代初,哈密顿路径问题被证明是属于“NP完备”的(NP—Complete)。我们无须探究NP完备性的理论,只指出下面这点就足够了:这一发现使得大多数理论计算机科学家们相信,哈密顿路径问题不可能存在有效的算法。不过,论明这一论断现在仍然是理论计算机科学中悬而未决的最重要问题之一,即所谓的“NP=P?”问题。[参看《科学》1984年9月号John E. Hopcroft所著“图灵机”一文。]这并不是说哈密顿路径问题不存在任何算法,只是说不存在有效的算法。例如,试考虑下面的算法:
给定一个有n个顶点的网络:
1、生成一组穿过网络的随机路径。
2、对于这一组路径中的每一条路径:
a、检查该路径是不是从起始顶点出发并到终止顶点结束如果不是,则从该组路径中去掉这一条路径。
b、检查该路径是否恰好经过n个顶点如果不是,则从该组路径中去掉这一条路径。
c、对于每个顶点,检查该路径是否经过该顶点。如果不是,则从该组路径中去掉这一条路径。
3、如果该组路径为非空集,则报告说存在一条哈密顿路径。如果该组路径为空集,则报告说不存在哈密顿路径。
这并不是一个完美的算法。但是,如果路径的生成足够随机,且所得的路径集合足够大,那么此算法给出正确答案的概率是相当高的。我在第一项DNA计算中所实现的正是这一个算法。
实验室中的7天
为了进行我的实验,我力图找到一个大小适当的哈密顿路径问题——小得很容易在实验室中解决,但又大得足以清楚地证明DNA计算的原理。我选择了图2左下方所示的有7个城市、14个航班的地图。一项非科学性的调查表明,找出这个网络中的唯一的哈密顿路径平均需要大约54秒(现在你可以开始了⋯⋯)。
为了简化我们的讨论,试考虑图2右上方的地图。这幅图中只有4个城市——亚特兰大、波士顿、芝加哥和底特律——由6个航班连接起来。问题是要确定是否存在一条以亚特兰大为起点而以底特律为终点的哈密顿路径。
作为第一步,我给每个城市分配一个随机的DNA序列。在我们的例子中,亚特兰大就成了ACCTGCAG,波士顿成了TCGGACTG等等。为了方便起见,可以设想其城市的DNA名字的前半部分为该城市的首名,而后半部分则是它的尾名。这样亚特兰大的尾名就是GCAG,而波士顿的首名就是TCGG。然后,我给每一个直达航班规定一个DNA“航班号”,即把航班起点城市的尾名和终点城市的首名连在一起充当该航班的航班号。在图2这个例子中,亚特兰大到波士顿的航班号为GCAGTCGG。
读者应当记得,每一股DNA都有它的Watson—Crick互补股。因此,每个城市都有其互补的DNA名字。例如,亚特兰大的互补名字就是TGAACGTC。
在作出这些编码后,我就让互补的DNA城市名和DNA航班号合成起来。(事实证明,DNA城市名本身基本上是不需要的。)我把这些不同的DNA序列的每一种都取一点(大约1O个分子),并把它们放入同一个试管中。为了开始计算,我只需加入水——再加上连接酶、盐以及其它几种成份以近似模拟细胞内的条件。总共只用了大约五十分之一茶匙的溶液。在1秒钟之内,哈密1顿路径问题的答案就被掌握在我的手中了。
为了了解这是怎么回事,我们来看看试管中发生了什么情况。例如,亚特兰大到波士顿的航班号(GCAGTCGG)和波士顿的互补名(AGCCTGAC)可能偶然地相遇。我们有意使得前一个序列以TCGG结尾,而后一个序列以AGCC开头。由于这两个序列是互补的,因此它们将结合在一起。如果所得的复合物接着又遇到了波士顿到芝加哥的航班号(ACTGGGCT),那么后者也将与这个复合物连接起来,因为前者的结尾(TGAC)与后者的开头(ACTG)是互补的。这样,随着DNA航班号被互补的DNA城市名不断地连接在一起,这些复合物就变得越来越长。然后,溶液中的连接酶将永久地把这些由DNA航班号组成的链连接起来。因此试管中就含有编码经过各个城市的随机路径的分子(正如算法第一步所要求的那样)。
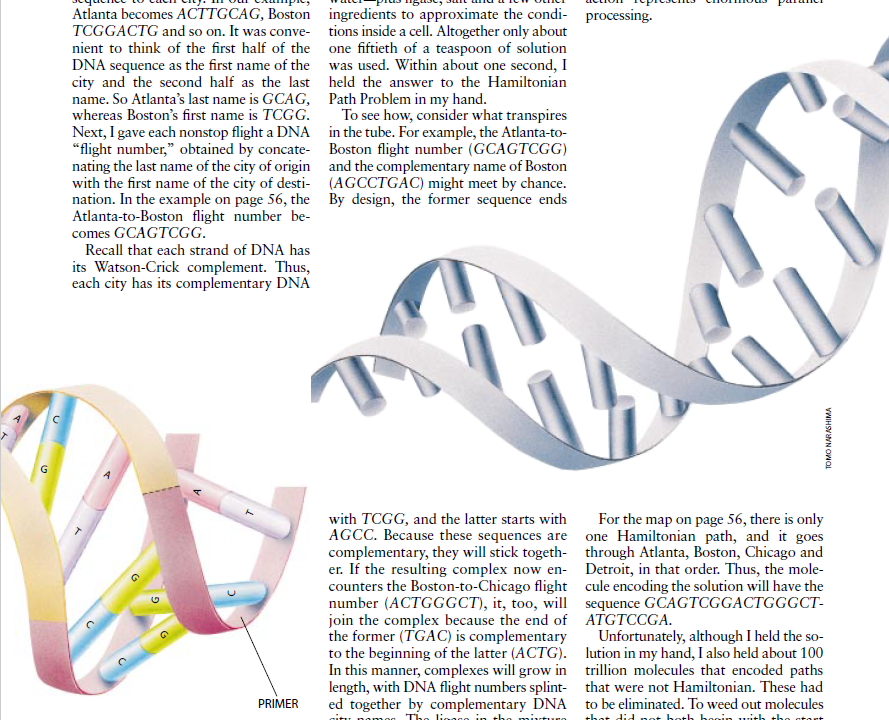
由于我在开始时使用了极其大量的分子,而本问题又只涉及少数几个城市,因此事实上可以肯定所形成的分子中至少有一种是属于编码哈密顿路径。一个数学问题的解答可以包含在一个分子中,想到这一点真是令人惊诧不已!
读者还应当注意到,所有这些路径都是由数百万亿个分子同时进行的相互作用一下子产生的。这一生物化学反应代表了规模极其巨大的并行处理过程。
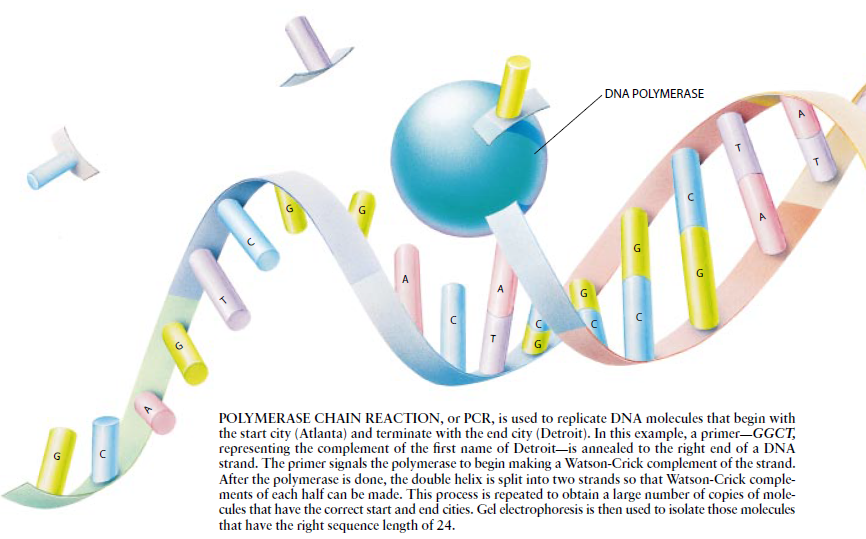
遗憾的是,尽管我手中已握有这个问题的解,但我手中同时还握有其它大约1百万亿个分子,它们编码的路径并不是哈密顿路径。这些分子必须除去。为了除去那些不是以起点城市开始或者不是以终点城市结束的分子序列,我采用了聚合酶链反应技术(PCR)。这一重要的技术需要两个短的DNA片段(启动子)的许多拷贝向DNA聚合酶发出信号,指示它开始进行Watson—Crick复制。我所用的启动子是起点城市的尾名(对亚特兰大为GCAG)和终点城市首名的Watson—Crick互补名(对底特律为GGCT)。这两个启动子协同工作:第一个启动子通知DNA聚合酶复制包含有正确的起点城市的DNA序列的互补序列,第二个启动子则启动编码正确的终点城市的DNA分子的复制。
聚合酶链反应是通过热循环过程进行的,即反复升高并降低试管中溶液的温度。温暖的条件促使DNA聚合酶开始复制,而较热的环境则使复制中所得的结合DNA股从其双螺旋结构分离,这样就可以对各个DNA片段再进行复制。
结果就是具有正确的起点城市和终点城市的DNA分子以指数增长的速度被复制出来。相反,那些只编码正确的起点城市但其终点城市却不正确、或者只编码正确的终点城市而其起点城市却不正确的DNA分子则以慢得多的线性速度复制。既无正确的起点城市又无正确的终点城市的DNA序列则根本不会被复制。这样,在聚合酶链反应完成之后,取少量的溶液,我就获得了含有正确的起点城市和正确的终点城市的分子的大量拷贝,但是不符合这一要求的分子则几乎没有。于是算法的第2a步就完成了。
接着我用凝胶电泳法来分离出那些具有正确长度(在图2的例子中,正确的长度为24)的分子。其它所有分子都被抛弃。这样就完成了算法的2b步。
为了检查剩下的DNA序列所编码的路径是否经过所有中间城市,我利用了一种所谓亲和性分离过程中的Watson—Crick退火结合。这一过程使用一个编码某城市(如波士顿)的互补名的DNA“探针”分子的多个拷贝。这些探针结合到一些显微铁球上(每一个铁球的直径大约为1微米)。
我让铁球悬浮在含有剩下的DNA分子的试管内,试管的条件有利于Watson—Crick配对过程的进行。只有那些含有正确的城市名(即波士顿)的DNA分子将退火结合到探针分子上。然后我把一块磁铁抵在试管壁上,把铁球都吸引到侧面来。我把试管中不含正确城市名的液相部分倒掉时,铁球就被保持在那里。
接着我向试管中加入新的溶剂并把磁铁移开;这样铁球又重新悬浮于溶液中。使溶液的温度升高,DNA分子就从探针上分离出来,重新溶解在液体中。然后我重新用磁铁把铁球再次吸引到试管的侧壁上,但这一次没有任何分子附着在铁球上。现在液体中含有所需要的DNA股(在本例中就是编码经过波士顿的路径的DNA分子),可以把它倒入一个新的试管中进行进一步的筛选。对于剩下的中间城市(本例中就是芝加哥)重复上述过程。这一反复进行的过程是整个实验中最繁琐的一部分,我在实验室中整整花了一天的功夫才完成它。
在亲和性分离结束时,算法的第2c步就完成了,而且我知道留在试管中的DNA分子应当正好就是编码哈密顿路径的分子。因此,只要试管中有DNA分子,我就可以断定该网络存在哈密顿路径。如果试管中没有DNA分子,那就意味着不存在这样的路径。幸运的是,为了作出这一判断,我可以再使用一步聚合酶链反应,跟着再进行一次凝胶电泳操作。我感到非常高兴的是,最终的分析表明,留在试管中的分子的确就是编码我们所要的哈密顿路径的分子。在实验室里度过7天之后第一次DNA计算终于完成了。
新领域诞生
未来的前景如何呢?很明显,分子计算机具有许多吸引人的性质。它们具有密度极大的信息存储能力。例如,一克DNA——在干的状态下其体积约为1立方厘米——可以存储的信息量相当于大约一万亿张CD的存储容量。而且它们具有极大规模的并行处理能力。即使是在只有五十分之一茶匙的溶液中进行的微型试验,也有大约10个DNA航班号在1秒的时间内被同时连接起来。我们不知道现有的最快的超级计算机能否如此迅速地完成这一样种任务。
分子计算机还可能具有异常之高的能量效率。现有计算机的效率要低得多,每焦耳最多只能执行1次操作。

世界各地的实验专家和理论科学家们正在努力进行研究以充分利用这些性质。他们能够成功地建造可与电子计算机分庭抗礼的分子计算机吗?这个问题的答案还有待观察。半个世纪以来人们对电子计算机的巨大财力和智力投入已经使电子计算机成了当代的奇迹——要动摇它的地位将是不容易的。
然而,如果仅是从这种实用的角度来看待有关DNA计算的研究,那未免太短视了。我的实验可以被看作是一个正在蓬勃兴起的科学新领域的一次展示,而我们正在迅速发展的驾驭分子世界的能力则使这个新领域的出现成为可能。这门新的分子科学的发展迹象比比皆是。例如,加利福尼亚州拉霍亚市斯克里普斯海洋学研究所的Gerald F.Joyce一代接一代地“繁殖”了数以万亿计的RNA分子,直至具有他所需要的催化性质的“优胜分子进化出来。[参看《科学》1993年4月号Gerald F.Joyce所著“定向的分子进化”一文。]麻省理工学院的Ju1ius Rebek创造出能够复制的分子,使我们得以了解地球上的生命可能是如何出现的。[参看《科学》1994年11月号Julius Rebek所著“合成的自复制分子”一文。]在关于DNA计算的研究的启发下,加利福尼亚理工学院的Erik Winfree合成出“智能分子复合物?”,这些复合物可以在“编程后把自己组装成具有任意复杂性的预定结构。还有其它许多例子。这一新领域的巨大潜力值得我们重视和培育。
对于我来说,只要知道用DNA进行计算是可能的就足够了。在过去半个世纪中,生物学和计算机科学都得到了极大的发展:几乎没有什么疑问的是,在新的一千年中它们将成为科学与经济进步的中心。但是生物学与计算机科学——生命与计算——是互相关联的。我深信,在这两门科学的接合处,还有许多伟大的发现在等待着那些苦苦寻找它们的人。
请 登录 发表评论