计算机模型揭示了水如何影响生物分子(如蛋白质)的结构与运动,从而为揭示它们的功能提供了线索。
在世界上的大多数地方,水即使不是免费的,也是非常便宜的。但是1986年夏天,Levitt(本文作者之一)为几乎不能打湿针头那么一点水花费了50万美元。这些钱用于支付在一台最先进的巨型超级计算机上将近两个星期的运行费用。其目的是要求开发水如何影响特定蛋白质的结构和运动的模型。
这种蛋白质就是在牛胰腺中发现的牛胰腺胰蛋白酶抑制剂(BPTI)。因为BPTI相对较小,所以比大多数其他蛋白质易于研究,所以成为计算机模型研制者的首选对象。以前,没有人把BPTI看成它实际上存在于活生生的细胞中,其周围环绕着数千个水分子。
结果证明50万美元花的值得,Levitt及其同事Ruth Sharon不仅发现以前的BPTI真空模型在预测蛋白质在真空世界中的形态和行为方面效果不好,而且这一发现有助于为计算化学家模拟真实水环境中的其他生物分子的结构铺平道路。
今天,由于计算技术的巨大进步,我们能在几天内在台式计算机上模拟蛋白质及其相关水分子,而费用仅为80美分电费。现在科学家已模拟了50多种蛋白质和核酸的水结构。
为什么了解水对生物分子结构的影响如此重要呢?从原则上讲,分子结构提供了它发挥功能的线索,有助于科学家揭开构成生命的生物化学相互作用的密码。了解水中生物分子的结构可能总有一天会有助于研究人员设计出抑制或促进各种生化途径的药物。
水的内部结构
为了解如何影响生物分子的结构,我们必须首先懂得水本身的独特性质。这些性质源于水的独特结构及这种结构允许水管理其它分子电荷的方式。
单个的水分子(H2O)具有独特的四面体几何形状,四面体中心有一个氧原子,氢原子占据4个角中的两个,而另两个角由负电荷云占据。负电荷云起源于氧和氢原子结构结合的方式。用简单的词语表述就是,氧有8个带负电荷的电子围绕其带正电荷的原子核:两个在内层,6个在外层。内层的最大容量为两个电子,因而它是饱和的:但外层却能容纳8个电子。氢只有一个电子。当氧与两个氢原子结合时,它就吸引每个氢的电子以填满其外层。因为每个氢原子的电子环绕氧原子花费的时间多于环绕自身带正电荷的原子核,因而水分子就具有了极性:两团带弱负电荷的电子云环绕氧原子,而两个氢原子则带弱正电荷。这样,两种类型的电荷互相抵销,水分子就呈中性。
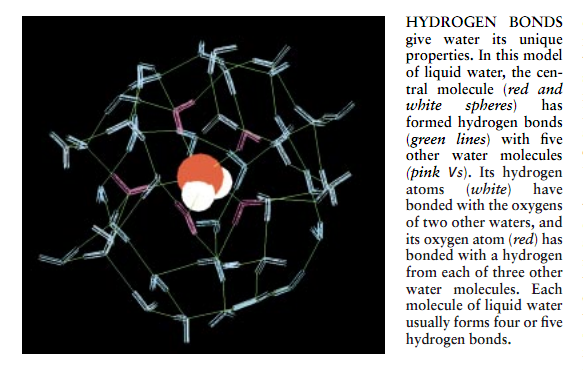
化学家通常不画环绕一个水分子中的氧原子的负电荷云,而是常常用v型描绘水(见图2)。V型的每边对应一个氢一氧键大约长10厘米。V型两边构成的角接近105度——略小于一个正四面体任意两边形成的109.5度角。
由于水分子具有极性,一个水分子带正电荷的氢原子同另一个水分子带负电荷的氧原子就最容易发生相互作用这种相互作用称为氢键。反映水的四面体几何结构,液态水的每个分子经常形成4个氢键:两个由其两个氢原子和另两个水分子的氧原子形成:两个由其氧原子和其它水分子的氢原子形成。但是,液态水一不同于泳(通常由水分子排列成正四面体几何形状的晶体组成)——的具体结构通常是十分随机和极不规则的。每个液态水分子的氢键实际数在3~6个之间,平均为4.5个。为了保持四面体性质,氢键结构使水具有开放性的、松散的结合结构:这不同于大多数其它液体(如袖或液态氮)的结构。
为了构建水的计算机模型,我们必须考虑两种不同的力:分子内的力和分子间的力。一个水分子内部的相互作用用短距离的弹力表示,它由每个分子内的氧原子和氢原子间的化学键产生。水分子间的相互作用用长距离的电力表示。分子内的力限制每个水分子的氧原子同其氢原子问键的长度一一以及这些键每条之间形成的角一只能取一定的值。这些力象弹簧一样作用,外力越压迫键,键越阻碍外力。
长距离的分子的力的表现不同于分子内的力:它们随着距离的增加而变小。从根本上讲,长距离力源于相反电荷间的相互吸引和相同电荷间的相互排斥。这些力既产生氢键又产生较弱的相互吸引,称为范德瓦耳斯力。
贝尔实验室的Aneesur Rahman和Frank H. Stillinger在60年代后期开创了计算机模拟水分子的先河。他俩在一个矩形盒中模拟216个水分子舳运动。(研究人员选择模拟216个水分子是因为构成盒子的长宽高都为6个分子那么长)在5微微(10秒的模拟中——这是使用当时的计算技术可能达到的最长模拟时间一—_Rahman和Stillinger发现,水的表现与水分子间的能量关系直接相关这种模拟能够提供水的许多特性(如一般结构、扩散率和汽化热)等的数量关系。
模拟生命
水在生命过程中的重要性不仅源于它能与其它水分子形成氢键,而且源于它能与不同类型的生物分子相互作用。由于水具有极性,水就易于与其它极性和带电分子如酸、盐糖蛋白质和DNA的各个区位进行相互作用。这种相互作用的结果使水分子能够溶解极性分子,这种分子因此被描述为亲水的(爱水)与此相反,水不能很好地与非极性分子如脂肪相互作用,其结果是出现水和油不能混和的现象。因此非极性分子被称为疏水的(“害怕水”)。
生物分子(如蛋白质和DNA)包含排列在长链中的亲水和疏水两部分。这些分子的三维结构由这种键折叠成更紧密构形的方式所支配,因而亲水部分在表面,它们能与水相互作用,而疏水部分则藏于内部,远离水。1959年Waiter Kaunann指出这种疏水效应对蛋白质的折叠而言是极为重要的:并且疏水性在蛋白质折叠中的作用今天依然是极有兴趣的课题(参见《科学》1991年5期“蛋白质迭叠问题”一文)。
构造水溶液中生物分子的计算机模型时必须考虑三种类型水:“有序的水”环绕着分子并与其相互作用,多余的“大量水”和其它任何可能位于分子内部的水。一个单一的细胞包含数十亿个水分子。未被生物分子原子占据的空间几乎充满了水分子。实际上,人体细胞绝大部分是水:人体重量中水约占60%。
我们怎样一起模拟生物分子的单个原子和这些类型的水呢?简单地说,我们首先要描述所有原子之间的基本相互作用关系.然后利用牛顿动力学来模拟这种系统的逐步演化。这种模拟需要两个基本的途径:一个是描述内部相互作用及水分子和生物分子之间的相互作用——换句话说就是分子内的力和分子间的力;一个是描述他,它们的随时间的运动情况,称为分子动力学。
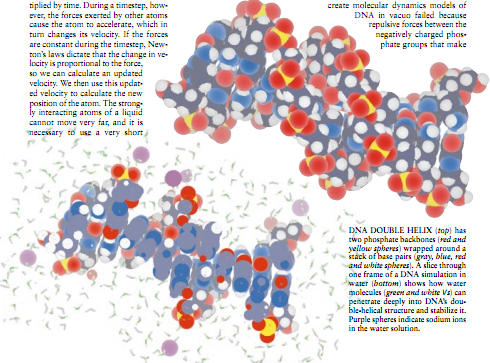
分子动力学产生了非常类似于电影中的画面的一系列造型。对每个院子运动情况的模拟是在一系列离散的时间区间上进行的,称为时步(timestep)。因此,一个原子的新位置就是原先的位置加上在给定的时步内移动的距离。如果没有外力作用于原子,其移动的距离就是先前位置的速度的函数,因为距离等于速度乘以时间。然而,在一个时步内.由于其它原子产生的力引起原子运动加速,从而改变了原子的速度。如果在一个时步期间,力是固定不变的.根据牛顿定律,速度与力成正比.这样我们就能算出最新的速度。然后利用最新的速度计算原子的新位置。液体中强烈相互作用的原子不可能运动得非常快,有必要使用非常短的时步:一飞秒(10-15秒)。在此时间内,一个水分子仅能移动其直径的1/50那么点距离。
在长距离的模拟中,计算有序水中生物分子的所有原子在每一时步中的变化产生了庞大的数据。例如,水中的一个小蛋白质在毫微秒(10-9)就产生50万组笛卡尔坐标数据,每个描述约10,000个原子的位置。电影采用这种模拟就极为清晰了。我们能看到每个水分子在数百万个画面上转动、移动和振动的情形。
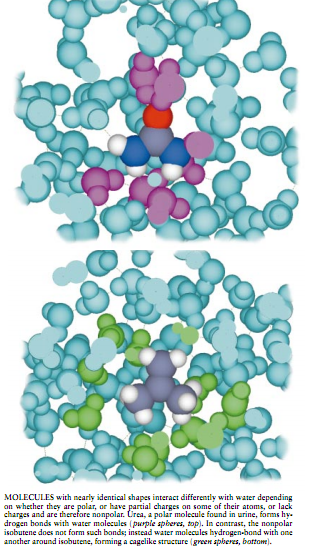
为描述计算机模拟如何描绘水影响分子动态的方式,让我们考虑两种简单的有机分子异丁烯和脲,它们形状相似,性质却完全不同。异丁烯是石油炼制业出产的一种燃料,是Y型、非极性(因而也是疏水的)分子,其主链包括4个碳原子,其中两个通过一个双键连接。脲是蛋白质代谢的产物,以尿的形式排出。它也具有Y型结构:一个羰基(C=O)连接两个氨基(NH2)。与异丁烯不同,脲是强烈的亲水极性分子。
我们在异丁烯和脲的分子动力学模拟时发现,环绕两种分子的水表现是不同的。水分子直接与脲相互作用,并与脲的氧原子和氢原子形成氢键,就像水分子相互之间那样。相反,水分子离开疏水的异丁烯,环绕分子的有序水形成笼状结构。
观察水分子如何与这些简单分子相互作用,有助于我们了解水与更复杂的生物分子如蛋白质和核酸间的表现。80年代后期Levitt和伦敦国立医学研究所的Miriam Hirshberg成功建立了包括水分子的DNA500微微秒模型;水分子与磷酸基一起形成氢键起到稳定双螺旋结构的作用。随后的含水DNA模拟表明,水分子几乎能够与DNA双螺旋的各个部分(包括构成遗传密码的碱基对)相互作用。
相反,水不能深入蛋白质内部结构,后者的疏水区域向内收缩成几近充满的实心。因此,蛋白质-水模拟重点是在蛋白质表面,此处没有内部结构那么紧密。
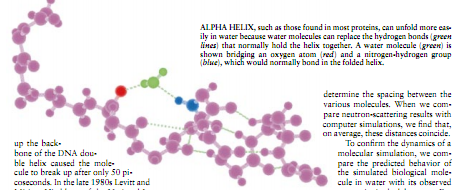
水分子引起了很有意思的几何形状——特别是在酶表面的深沟中,这些酶促进细胞中的化学反应。氢键水分子难于填充这些沟,并容易被配位体取代——配位体是酶将与之相互作用的分子——这也许能解释为什么酶的活性部位常常发生在沟中。我们常常发现空活性部位的水分子的排列模仿实际配位体的形状和结构,这种知识有时用于药物的设计。
真实世界中的生命
水中生物分子模拟多大程度上接近现实情况呢?遗憾的是,我们还不能确切地回答这个问题,因为还没有实验技术能够像计算机模拟一样提供各个分子及其相互作用的详尽信息。我们所能做到的事情就是将模拟的各种总结果和平均值与实验结果进行比较。
能用来检验水中模拟分子结构的一种最重要的方法就是中子和X线散射。在中子散射中,我们向少许试样散射中子的情况。分子间的每一间距就是微小的孔洞,并将产生特征衍射类型。通过分析这些类型,我们就能轻松的确定各种分子间的距离。将中子散射结果与计算机模拟结果进行比较时,我们发现,从平均值上看,其距离是吻合的。
为了验证分子模拟动力学,我们比较了水分子模拟生物分子的预测行为表现及实验室中观察到的特性。例如,大多数蛋白质至少包含了一条阿尔法螺旋,其氨基酸组成蛋白质螺旋从而形成旋管。我们从实验中得知,热能引起这些阿尔法螺旋模拟尝试中,随着温度的升高,螺旋却保持不变。只有在模型中加入这一因素,它才接近阿尔法螺旋的实际表现。Levitt和华盛顿大学的Valerie Daggett进行了这种模拟。
这类计算机模拟产生了越来越多的有关生命体中各种生物分子及其功能表现的信息。然而,在试图进行水环境中更加复杂的生物分子模拟时,依然面临着计算技术的限制和超级计算机运行时间费用的问题。科学家在杂志上发表生物分子模型时,他们通常将模型在素色黑色背景下绘成鲜明的图形。现在我们知道,这些分子存在的背景——水——也是同样重要的。
请 登录 发表评论